Análise exploratória de casos de Dengue notificado pelo SUS
Neste tutorial vamos explorar como começar a entender a descrição de casos do sis tema de notificação de agravos do SUS, o SINAN. Estes dados são bastante ricos e a documentação sobre o significado de cada uma das variáveis pode ser encontrada aqui.
[1]:
from pysus.ftp.databases.sinan import SINAN
import pandas as pd
import geopandas as gpd
%pylab inline
sinan = SINAN().load()
%pylab is deprecated, use %matplotlib inline and import the required libraries.
Populating the interactive namespace from numpy and matplotlib
Primeiro vamos começar pelo carregamento dos dados a partir do servidor do DataSUS. Como o dado está no formato Parquet, nós vamos utilizar a biblioteca pandas para visualizar os dados em um Dataframe:
[2]:
cols = [
'ID_AGRAVO', 'DT_NOTIFIC', 'SEM_NOT', 'NU_ANO', 'ID_MUNICIP',
'DT_SIN_PRI', 'SEM_PRI', 'NU_IDADE_N', 'CS_SEXO', 'CS_GESTANT',
'latitude', 'longitude',
'NM_DISEASE'
]
casos = sinan.download(sinan.get_files('DENG', 2010)).to_dataframe()
100%|████████████████████████████████████████████████████████████████| 30.5M/30.5M [00:00<00:00, 6.93GB/s]
[3]:
munrj = gpd.read_file('./rj-municipalities.json')
munrj.plot();
/home/bida/micromamba/envs/pysus/lib/python3.11/site-packages/geopandas/array.py:93: ShapelyDeprecationWarning: __len__ for multi-part geometries is deprecated and will be removed in Shapely 2.0. Check the length of the `geoms` property instead to get the number of parts of a multi-part geometry.
aout[:] = out
/home/bida/micromamba/envs/pysus/lib/python3.11/site-packages/geopandas/plotting.py:33: ShapelyDeprecationWarning: Iteration over multi-part geometries is deprecated and will be removed in Shapely 2.0. Use the `geoms` property to access the constituent parts of a multi-part geometry.
for poly in geom:
/home/bida/micromamba/envs/pysus/lib/python3.11/site-packages/descartes/patch.py:62: ShapelyDeprecationWarning: The array interface is deprecated and will no longer work in Shapely 2.0. Convert the '.coords' to a numpy array instead.
vertices = concatenate([
[4]:
casos.head()
[4]:
| TP_NOT | ID_AGRAVO | DT_NOTIFIC | SEM_NOT | NU_ANO | SG_UF_NOT | ID_MUNICIP | ID_REGIONA | ID_UNIDADE | DT_SIN_PRI | ... | PLASMATICO | EVIDENCIA | PLAQ_MENOR | CON_FHD | COMPLICA | HOSPITALIZ | DT_INTERNA | UF | MUNICIPIO | TP_SISTEMA | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 2 | A90 | 2010-04-14 | 201015 | 2010 | 35 | 352510 | 1348 | 2038641 | 2010-04-07 | ... | 9 | 1 | ||||||||
| 1 | 2 | A90 | 2010-04-15 | 201015 | 2010 | 35 | 352510 | 1348 | 2038641 | 2010-04-09 | ... | 9 | 1 | ||||||||
| 2 | 2 | A90 | 2010-04-13 | 201015 | 2010 | 35 | 352510 | 1348 | 2038641 | 2010-04-09 | ... | 9 | 1 | ||||||||
| 3 | 2 | A90 | 2010-04-13 | 201015 | 2010 | 35 | 352510 | 1348 | 2038641 | 2010-04-08 | ... | 9 | 1 | ||||||||
| 4 | 2 | A90 | 2010-04-20 | 201016 | 2010 | 35 | 352510 | 1348 | 2038641 | 2010-04-15 | ... | 1 |
5 rows × 66 columns
Estes dados correspondem a todos os casos de dengue notificado ao SUS durante um período. Neste caso de 2015 a 2016. Para podermos tratar adequadamente estes dados para fins de visualização ou análise precisamos corrigir os tipos das colunas. Por exemplo vamos converter as datas.
[5]:
casos.DT_NOTIFIC = pd.to_datetime(casos.DT_NOTIFIC)
casos.DT_SIN_PRI = pd.to_datetime(casos.DT_SIN_PRI)
Para poder organizar os dados temporalmente, é útil indexar a tabela por alguma variável temporal
[6]:
casos = casos.set_index('DT_NOTIFIC')
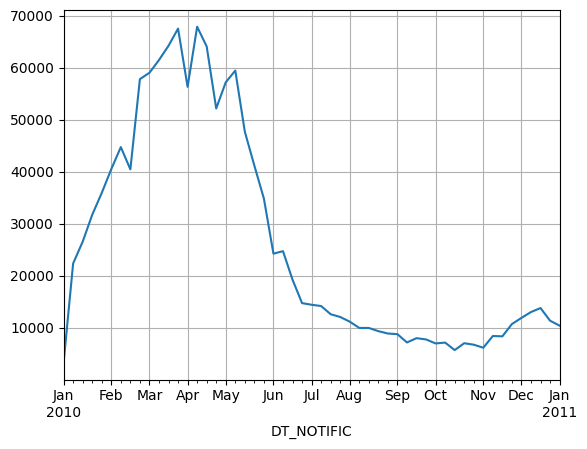
Agora podemos plotar o número de casos por semana de dengue na cidade do Rio de Janeiro.
[7]:
casos.ID_AGRAVO.resample('1W').count().plot(grid=True);
Suponhamos agora que desejamos visualizar em um mapa os casos que ocorreram, por exemplo entre janeiro e março de 2015